10X Genomics 3′ 技术
1、技术优势
通量高:一次可以同时测8个样本,每个样本最多可以检测到上万个细胞。
周期短:7分钟内完成上万个细胞封装,一天之内完成细胞悬液制备、单细胞捕获、扩增以及建库。
单细胞率高:单个GEMs捕获到多个细胞的概率极低(<0.9%/1000cells)。
捕获效率高:细胞捕获效率高达65%。
应用范围广:动物细胞和植物细胞均可以进行单细胞测序,已广泛应用于肿瘤细胞异质性、免疫细胞群体检测和胚胎发育研究等。
2、实验操作方法及流程
如图所示,10X Genomics的Chromium系统利用微流控系统将含有Barcode的凝胶珠(Gel Beads)、细胞和酶的混合物、油三者混合,形成GEMs(油包水的微体系)。GEMs形成后,细胞在其中裂解,释放出mRNA,凝胶珠自动溶解释放大量barcode序列,利用PloyT引物捕获液滴中的mRNA。随后mRNA逆转录产生带有Barcode和UMI信息的cDNA,构建标准测序文库。

2.1 单细胞悬液制备
新鲜组织样本需要消化成单细胞悬液,对于培养的细胞或已经处于悬浮状态的细胞,需要对细胞洗涤以去除培养基。在进行单细胞RNA-seq之前,可以使用冷冻保存或甲醇固定来储存细胞。对于进行单细胞测序实验的样本,要求细胞活性>70%, 细胞浓度在700-1200细胞/μl,细胞尺寸最大不超过30μm。
2.2 细胞分选
如上图所示,10X Genomics运用微流控系统进行细胞分选,凝胶珠匀速地从左方(图示)进入,待分选的细胞和酶从下方(图示)以一定时间间隔进入,与凝胶珠一起油相中形成GEMs(Gel Bead in emulsion)。如图所示,凝胶珠上的寡核苷酸链依次为Illumina TruSeq Read 1测序引物、16bp 10X Barcode序列(用于区分细胞)、12 bp unique molecular identifier(UMI,区分细胞的不同转录本并去除PCR Duplications,降低扩增偏好性)、30 bp poly dT反转录引物。在随后的文库构建中,相同的细胞扩增出的cDNA带有相同的10x Barcode,其中来自相同的转录本的cDNA会带有相同的UMI,由此,便可区分出不同细胞扩增出的cDNA。一般而言,每个GEMs只会包含一个细胞和一个凝胶珠,但有时候也会出现一个GEM里没有或有多个细胞的情况,对于这种GEMs则需要通过在后续分析中识别出来并进行去除。

2.3 cDNA生成与扩增
形成GEMs后,凝胶珠溶解,细胞裂解释放mRNA,凝胶珠中的polyT捕获mRNA中的polyA,并逆转录生成用于测序的cDNA。然后破坏油包水结构,使用磁珠纯化cDNA并进行清洗,再进行扩增反应,纯化后进行质检,质检合格后进行文库构建。


2.4 文库构建
cDNA扩增完成后,利用酶将cDNA打断成片段,通过末端修复、并加入A碱基,连接Read2测序引物,并通过PCR扩增引入样品Index、P5、P7接头,最后进行片段筛选,从而构建含有P5和P7接头的cDNA文库,文库质检合格后再上机测序。

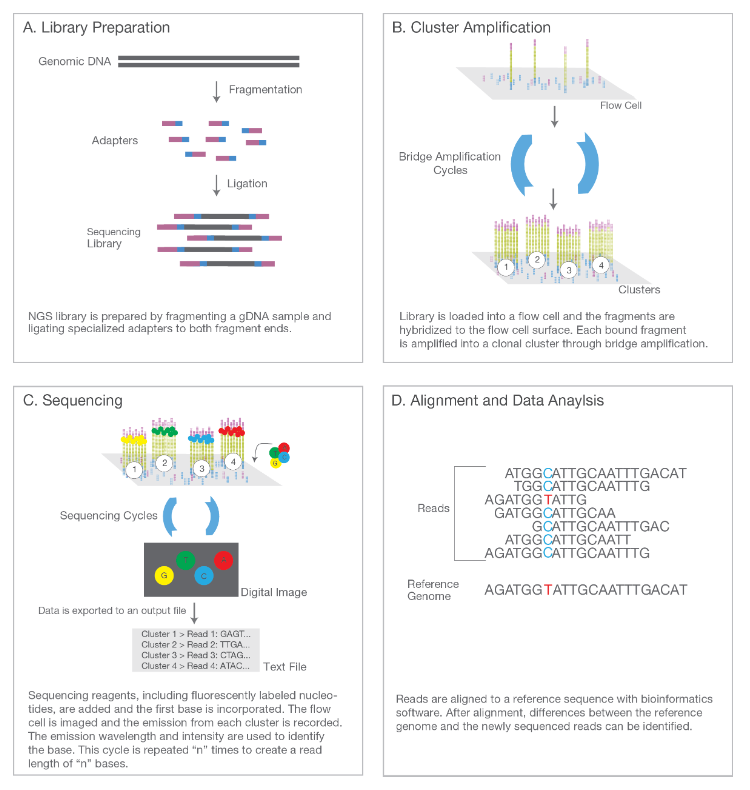
2.5 文库测序
文库质检合格后,就使用Illumina双端测序平台进行测序。如图所示,Illumina测序的核心在于利用可逆终止的、荧光标记的dNTP进行边合成边测序(Sequencing-By-Synthesis,SBS)。